JuliaとPythonで擬似コードや数式を動かそうとしたときの比較
この記事はJulia Advent Calendar 2017の17日目の記事です.
普段はpythonばかり書いていて,juliaは最近文法覚えてきたかなレベルなので色々許してください.
コードの全体はここにあります.
概要
- この記事では擬似コードや数式を可能な限りそのままプログラムすることを目的とします.
- unicode文字を使いまくって以下の画像のようなプログラムを作成します.
- juliaとpythonで実装して,書きやすさと実行速度を比較します.
- 書きやすさが悪化するので型指定はしません.
- 結論は以下です.
- juliaのほうが色んなunicode文字が使えるから,書きやすく可読性が高い.
- インデックスが1から始まるのがいい.
- juliaのほうが倍程度速くなることもあるけど,思ったより速くならない (型指定してないから)
- juliaのeinsumを何も考えずに使うと激遅になる.(僕がやらかしてるかも)
unicode文字は以下の埋め込みではおそらく微妙な感じに表示されますが,ちゃんと設定されたエディタやjupyter notebookでは以下のように表示されます.
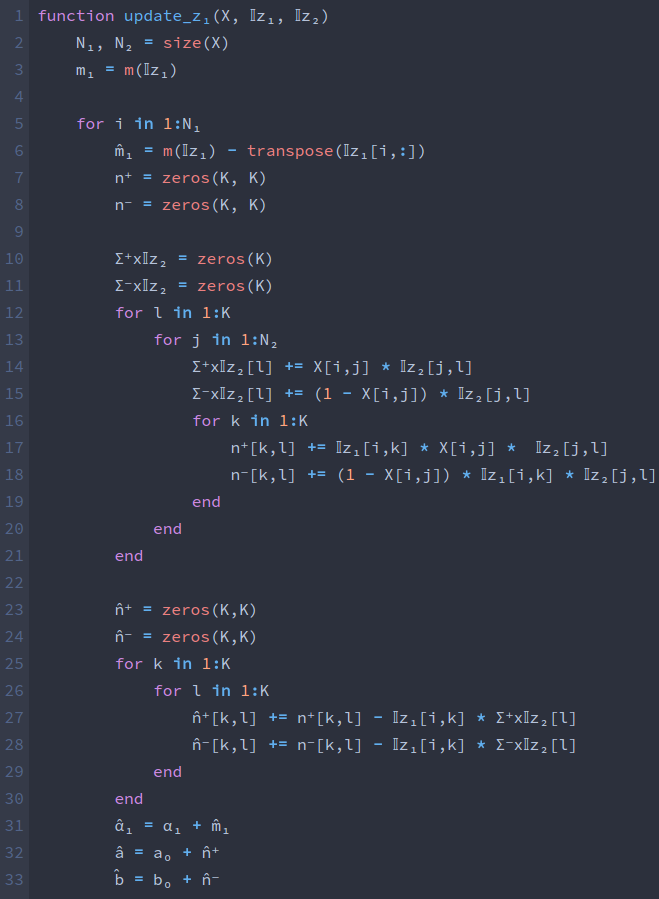
Table of Contents
はじめに (式or擬似コードに可能な限り近いプログラム)
数式を見て実装するときや論文に書いてある擬似コードを実装するとき,可能な限りそれらに近いプログラムを書くようにしている.
数式/擬似コードとプログラムを近づけるために,などunicode文字を多用する.
このようなプログラムを書くことで,
という利点がある.
例えばNo-U-Turn Sampler Hoffman+, 2011の擬似コード(の一部,論文より引用)は

これに対して書いたpythonコードは以下.
def BuildTree(θ, r, u, v, j, ε): if j == 0: θd, rd = Leapfrog(x, θ, r, v * ε) if np.log(u) <= (L(*θd) - 0.5 * np.dot(rd, rd)): Cd_ = [[θd, rd]] else: Cd_ = [] sd = int(np.log(u) < (Δ_max + L(*θd) - 0.5 * np.dot(rd, rd))) return θd, rd, θd, rd, Cd_, sd else: θ_minus, r_minus, θ_plus, r_plus, Cd_, sd = BuildTree(θ, r, u, v, j - 1, ε) if v == -1: θ_minus, r_minus, _, _, Cdd_, sdd = BuildTree(θ_minus, r_minus, u, v, j - 1, ε) else: _, _, θ_plus, r_plus, Cdd_, sdd = BuildTree(θ_plus, r_plus, u, v, j - 1, ε) sd = sdd * sd * int((np.dot(θ_plus - θ_minus, r_minus) >= 0) and (np.dot(θ_plus - θ_minus, r_plus) >= 0)) Cd_.extend(Cdd_) return θ_minus, r_minus, θ_plus, r_plus, Cd_, sd
pythonではギリシャ文字などの一部のunicode文字を変数に使うことができる.
しかし,一部の例えば
や
などの記号は使うことができないため,微妙に見難い感じになってしまっていた.
探してみるとjuliaで同じことをしている人がいて,こちらのほうがだいぶ良さそうだった.
juliaでは↑であげたような文字に加えて
みたいな修飾文字や不等号の
とかを使える.
juliaで使えるunicode文字一覧はこちら.
juliaすごい.ということで,以下ではこれまでpythonで実装したコードをjuliaで書きなおし,書きやすさと実行速度を比較する.
NUTSは既にやられているようなので
- Dynamic Time Warping
- Stochastic Block Model
について実装する.
juliaは型をちゃんと指定するともっと速くなるが,「そのまま実装する」という目的に反するのでやらない.
ちなみにNUTSのpython実装の話はこっち.
Dynamic Time Warping
Dynamic Time Warping (DTW) は,2つの連続値系列データのいい感じの距離を求めるアルゴリズム. 動的計画法で解く. pythonで実装したときの記事はこっち ksknw.hatenablog.com
A global averaging method for dynamic time warping, with applications to clustering (DTW自体の論文ではない) の擬似コードを参考にしてプログラムを書く. 擬似コードは以下.


(ただしこの擬似コードは多分間違っているので,m[i,j]の遷移前のインデックスをsecondに入れるように変えた.)
python
δ = lambda a,b: (a - b)**2 first = lambda x: x[0] second = lambda x: x[1] def minVal(v1, v2, v3): if first(v1) <= min(first(v2), first(v3)): return v1, 0 elif first(v2) <= first(v3): return v2, 1 else: return v3, 2 def dtw(A, B): S = len(A) T = len(B) m = [[0 for j in range(T)] for i in range(S)] m[0][0] = (δ(A[0],B[0]), (-1,-1)) for i in range(1,S): m[i][0] = (m[i-1][0][0] + δ(A[i], B[0]), (i-1,0)) for j in range(1,T): m[0][j] = (m[0][j-1][0] + δ(A[0], B[j]), (0,j-1)) for i in range(1,S): for j in range(1,T): minimum, index = minVal(m[i-1][j], m[i][j-1], m[i-1][j-1]) indexes = [(i-1,j), (i,j-1), (i-1,j-1)] m[i][j] = (first(minimum)+δ(A[i], B[j]), indexes[index]) return m
擬似コードや数式ではインデックスを1から始めることが多いが,pythonは0からなので,頭の中でインデックスをずらしながらプログラムを書く必要がある. 他にはv1が添字っぽくない程度で,大体そのまま書けた.
julia
δ(a,b) = (a - b)^2 # first(x) = x[1] firstは元からあるのでいらない second(x) = x[2] function minVal(v₁, v₂, v₃) if first(v₁) ≤ minimum([first(v₂), first(v₃)]) return v₁, 1 elseif first(v₂) ≤ first(v₃) return v₂, 2 else return v₃, 3 end end function DTW(A, B) S = length(A) T = length(B) m = Matrix(S, T) m[1, 1] = [δ(A[1], B[1]), (0,0)] for i in 2:S m[i,1] = [m[i-1, 1][1] + δ(A[i], B[1]), [i-1, 1]] end for j in 2:T m[1,j] = [m[1, j-1][1] + δ(A[1], B[j]), [1, j-1]] end for i in 2:S for j in 2:T min, index = minVal(m[i-1,j], m[i,j-1], m[i-1,j-1]) indexes = [[i-1, j], [i, j-1], [i-1, j-1]] m[i,j] = first(min) + δ(A[i],B[j]), indexes[index] end end return m end
endがある分pythonより長い.一方でpythonでは使えない
とか
が使えるので,より忠実な感じになっている.
実際に書いてみるとわかるけど,インデックスが擬似コードと同じなのは結構大事で,全然頭を使わずにそのまま書けた.
実行速度比較
juliaはpythonに比べて速いらしいので実行速度を比較した. シェルのtime を使って実行速度を比較した. コードの全体はここにある.
julia dtw_julia.jl 2.62s user 0.30s system 110% cpu 2.641 total python dtw_python.py 2.76s user 0.11s system 99% cpu 2.873 total
期待していたよりも全然速くならなかった. 実行時間が短くてコンパイルのオーバヘッドが大きいのかなと思ったから,forで10回実行するようにした結果が以下.
julia dtw_julia.jl 21.97s user 0.66s system 101% cpu 22.355 total python dtw_python.py 25.81s user 0.78s system 99% cpu 26.591 total
多少速い気がするけど,期待としては数十倍とかだったので,いまいち. 型指定していないとこれぐらいなのかな(?)
Stochastic Block Model
Stochastic Block Model (SBM)は非対称関係データのクラスタリング手法.
隠れ変数の事後分布をサンプリングして近似する(周辺化ギブスサンプラー).
今回の例では特にクラスタ割り当て
の事後分布を求めて,サンプリングする部分をやる.
pythonでの実装したときの記事はこっち. ksknw.hatenablog.com
関係データ学習という本に載っているクラスタzの事後確率に関する数式は以下.
,
をサンプリングする。
他の変数がgivenだとした時の、の事後確率は、
ここで、
はガンマ関数で、
はパラメータ
この事後分布を求めてサンプリングする部分をpythonとjuliaで比較する. 全部書くと見づらいので一部だけ,コードの全体は同じくここにある.
python
n_pos = np.einsum("ikjl, ij", np.tensordot(z1, z2, axes=0), X) # n_pos_kl = n_pos[k][l] n_neg = np.einsum("ikjl, ij", np.tensordot(z1, z2, axes=0), 1 - X) m1_hat = lambda i: m1 - z1[i] # m1_hat_k = m1_hat[k] n_pos_hat = lambda i: n_pos - np.einsum("kjl, j", np.tensordot(z1, z2, axes=0)[i], X[i]) n_neg_hat = lambda i: n_neg - np.einsum("kjl, j", np.tensordot(z1, z2, axes=0)[i], 1 - X[i]) α_1_hat = lambda i: α1 + m1_hat(i) a_hat = lambda i: a0 + n_pos_hat(i) b_hat = lambda i: b0 + n_neg_hat(i) aᵢhat = a_hat(i) bᵢhat = b_hat(i) p_z1ᵢ_left = logΓ(aᵢhat + bᵢhat) - logΓ(aᵢhat) - logΓ(bᵢhat) p_z1ᵢ_right_upper = logΓ(aᵢhat + np.dot(X[i], z2)) + logΓ(bᵢhat + np.dot((1 - X[i]), z2)) p_z1ᵢ_right_lower = logΓ(aᵢhat + bᵢhat + m2) p_z1ᵢ = (α_1_hat(i) * exp(p_z1ᵢ_left + p_z1ᵢ_right_upper - p_z1ᵢ_right_lower)).prod(axis=1) p_z1ᵢ = p_z1ᵢ.real p_z1ᵢ = p_z1ᵢ / p_z1ᵢ.sum() new_z1.append(onehot(choice(range(nb_k), p=p_z1ᵢ), nb_k))
数式には
や
などが頻出するが,pythonではこれらの文字を使うことができない.
このため,m1_hatやn_posなどの変数名になってしまっている.
julia
@einsum n⁺[k,l] := X[i,j] * 𝕀z₁[i,k] * 𝕀z₂[j,l] @einsum n⁻[k,l] := (ones(X)[i,j] - X[i,j]) * 𝕀z₁[i,k] * 𝕀z₂[j,l] m̂₁ = m(𝕀z₁) - transpose(𝕀z₁[i,:]) @einsum Σ⁺x𝕀z₂[i,l] := X[i,j] * 𝕀z₂[j,l] @einsum Σ⁻x𝕀z₂[i,l] := (ones(X)[i,j] - X[i,j]) * 𝕀z₂[j,l] @einsum n̂⁺[k,l] := n⁺[k,l] - 𝕀z₁[i,k] * Σ⁺x𝕀z₂[i,l] @einsum n̂⁻[k,l] := n⁻[k,l] - 𝕀z₁[i,k] * Σ⁻x𝕀z₂[i,l] α̂₁ = α₁ + m̂₁ â = a₀ + n̂⁺ b̂ = b₀ + n̂⁻ temp⁺ = zeros(â) temp⁻ = zeros(â) temp = zeros(â) for j in 1:size(temp⁺)[1] temp⁺[j,:] = Σ⁺x𝕀z₂[i,:] temp⁻[j,:] = Σ⁻x𝕀z₂[i,:] temp[j,:] = sum(𝕀z₂,1) end @einsum p_z₁[k,l] := exp(logΓ(â + b̂)-logΓ(â)-logΓ(b̂) + logΓ(â+temp⁺)+logΓ(b̂+temp⁻)-logΓ(â+b̂+temp))[k,l] p_z₁ = α̂₁ .* transpose(prod(p_z₁, 2)) p_z₁ /= sum(p_z₁) 𝕀z₁[i,:] = onehot(sample(1:K, Weights(p_z₁[:])), K)
pythonに対してjuliaではα̂₁やn̂⁺などをそのまま変数名に使えて便利.
実行速度比較
python sbm_python.py 8.74s user 0.09s system 354% cpu 2.492 total julia sbm_julia.jl たぶん60000sぐらい(終わらない...)
(なにか間違っているかもしれないが,) なんとjuliaのほうが圧倒的に遅くなってしまった. というか同じ量の計算をさせると終わらない... 調べてみるとeinsumがだめっぽいので,以下ではforで書き直す. (とはいえさすがに遅すぎるので何かやらかしている気がする.)
einsumをfor文で書き直す
juliaのマクロ(@のやつ)はコンパイル時に別の式に展開されるらしい. einsumのマクロもそれぞれの@einsumに対して多重のfor文を展開しているらしく,しかも単独でもnumpyのeinsumより遅いこともあるらしい.
ということで,普通にfor文による実装に変更して,実行速度を比較し直す. コードは以下.
m̂₁ = m(𝕀z₁) - transpose(𝕀z₁[i,:]) n⁺ = zeros(K, K) n⁻ = zeros(K, K) Σ⁺x𝕀z₂ = zeros(K) Σ⁻x𝕀z₂ = zeros(K) for l in 1:K for j in 1:N₂ Σ⁺x𝕀z₂[l] += X[i,j] * 𝕀z₂[j,l] Σ⁻x𝕀z₂[l] += (1 - X[i,j]) * 𝕀z₂[j,l] for k in 1:K n⁺[k,l] += 𝕀z₁[i,k] * X[i,j] * 𝕀z₂[j,l] n⁻[k,l] += (1 - X[i,j]) * 𝕀z₁[i,k] * 𝕀z₂[j,l] end end end n̂⁺ = zeros(K,K) n̂⁻ = zeros(K,K) for k in 1:K for l in 1:K n̂⁺[k,l] += n⁺[k,l] - 𝕀z₁[i,k] * Σ⁺x𝕀z₂[l] n̂⁻[k,l] += n⁻[k,l] - 𝕀z₁[i,k] * Σ⁻x𝕀z₂[l] end end α̂₁ = α₁ + m̂₁ â = a₀ + n̂⁺ b̂ = b₀ + n̂⁻ p_z₁ = α̂₁ for k in 1:K for l in 1:K p_z₁[k] *= exp(logΓ(â[k,l] + b̂[k,l])-logΓ(â[k,l])-logΓ(b̂[k,l]) \ + logΓ(â[k,l]+Σ⁺x𝕀z₂[l])+logΓ(b̂[k,l]+Σ⁻x𝕀z₂[l])-logΓ(â[k,l]+b̂[k,l]+sum(𝕀z₂,1)[l])) end end p_z₁ /= sum(p_z₁) 𝕀z₁[i,:] = onehot(sample(1:K, Weights(p_z₁[:])), K)
for...endのせいで見難く感じるが,これは普段pythonばかり書いていてfor文に拒絶反応があるからかもしれない. 書いている時の感想としては,行列の向きとか気にしなくていいので,einsumと同じ程度には書きやすい. 実際にfor文を全部とっぱらえば数式と近くなるはず.
実行速度比較
python sbm_python.py 8.74s user 0.09s system 354% cpu 2.492 total julia sbm_julia_2.jl 3.62s user 0.46s system 114% cpu 3.559 total
無事にpythonの半分以下の時間で処理が終わるようになった. 普段python使っていると無意識的にfor文を避けていたのが, juliaなら何も気にせずに普通にfor文書きまくれば良さそう.
おわりに
DTWとSBMという2つのアルゴリズムについて,pythonとjuliaでの実装を比較した.
| 書きやすさの改善 | 実行速度の改善 | |
|---|---|---|
| DTW | インデックスが1から.数字の添字 | 25.81s → 21.97s |
| SBM (einsum) | α̂₁やn̂⁺をそのまま書ける | 激遅 (やらかしているかも) |
| SBM (for) | ↑ | 8.74s → 3.62s |
pythonをjuliaで書き直すことで
- 数式の添字をそのまま書くことができ,書きやすさと可読性が向上した.
- 素直にfor文を使えば実行速度は改善した.
- juliaのeinsumは激遅だった.(僕がやらかしてるかも)
- 型の指定をしていないからか,実行速度の改善は限定的だった.
また,やっていて気づいたこととして
- jupyter notebook上で実行しつつ書くというやり方だと,コンパイル時間が以外と鬱陶しい
- a\hat はOKだけど,\hat aはだめとか.\leはOKだけど,\leqqはだめとかunicode関連で微妙につまることがあった.
この記事ではやらなかったこととして以下がある.気が向いたらやろうと思っている.
はてなだと表示が微妙だけど,jupyter とかでは綺麗に表示されて見やすいよ!(大事なこと)
追記 (2017/12/17)
Qiitaにて,juliaのコードについてのコメントをいただきました.ありがとうございます.
- DTWについて,こちらに書かれているように(Matrixの要素の型推論とベクトルの代わりにタプルを使用)コードを変更すると,数十倍程度高速化します.
- SBMについて,グローバル変数になっているパラメータにconstを指定するだけで,実行時間が90%程度になります.
もう少し自分でも高速化のために,あれこれやってみたいと思います.
参考
- Julia Advent Calendar 2017 - Qiita
- [1111.4246] The No-U-Turn Sampler: Adaptively Setting Path Lengths in Hamiltonian Monte Carlo
- Unicodeプログラミングのすゝめ - りんごがでている
- Unicode Input · The Julia Language
- DTW (動的時間伸縮法)の実装 - やったことの説明
- pythonでNUTSの実装 - やったことの説明
- A global averaging method for dynamic time warping, with applications to clustering
- 関係データ学習の実装 PythonでStochastic Block Modelの実装 - やったことの説明
- 関係データ学習 | 書籍情報 | 株式会社 講談社サイエンティフィク
- Julia Language Programming
- Performance · Issue #1 · ahwillia/Einsum.jl · GitHub
pytorchで Canonical Correlation Analysis (正準相関分析)の実装
はじめに
- pytorchの練習も兼ねて,Canonical Correlation Analysis (正準相関分析)をpytorchを使って実装する.
- 本当は分散共分散行列からなる行列の一般化固有値問題を解くが,今回は勾配法で解を求める.
- pytorchのプログラムが間違っていないことを確認するためにscikit-learnでもやる.
Canonical Correlation Analysis (正準相関分析)
こちらの資料を参考にプログラムを書く.
主成分分析が多次元の値に対して,分散が大きい方向に射影するアルゴリズムなのに対して,正準相関分析では2つの多次元変数を射影先で相関が大きくなるように射影するアルゴリズムである.
多次元のデータ
と
間の正準相関を考える.
ここで
はデータ数(系列データのときはデータ長),
は
の次元を表す.
の射影ベクトルをそれぞれ
,
とする.
の平均がそれぞれ0のとき,射影後の分散
と共分散
は,
,
,
.
よって,射影後の相関は
になる.
ここで,
とか
を単純に
倍しても相関は変化しない.
最適化のときに解が複数あるのは面倒なので,制約条件
を加える.
以上から以下を解くことで正準相関が求まる.
これはラグランジュの未定乗数法で解くことができて,一般化固有値問題になるが,今回はこの式のまま勾配法で解く.
生成データでのテスト
以下のように適当に生成したデータを使って,正準相関分析のプログラムをテストする.
以下では
の3つの信号を使う.
はもともと同じ信号に大きめのノイズが乗ったもの.dummyは適当なノイズだけのもの.
間では正準相関の値が大きくなり,
,dummy間では正準相関の値が小さくなってほしい.
import pylab as plt import numpy as np import torch from torch.autograd import Variable import logging import coloredlogs from tqdm import tqdm logger = logging.getLogger(__name__) coloredlogs.install( #fmt="%(asctime)s %(levelname)s %(message)s" fmt="%(levelname)s %(message)s" ) %matplotlib inline logger.setLevel("DEBUG") if logger.level >= logging.WARNING: tqdm = lambda x:x
u1 = np.r_[np.zeros(40), np.ones(30), np.ones(50)*2, np.ones(30), np.zeros(20), np.ones(30) ] u2 = np.r_[np.ones(25), np.zeros(25), np.ones(25)*2, np.ones(55), np.zeros(35), np.ones(35) ] u = np.c_[u1, u2]
plt.plot(u) plt.show()

sx = np.random.random(len(u)) * 10 sy = np.random.random(len(u)) * 10 sd = np.random.random(len(u)) * 10
x = np.c_[
u1 + sx + np.random.random(len(u)),
u2 - sx + np.random.random(len(u)),
]
plt.plot(x)
plt.show()

y = np.c_[
u1 + sy + np.random.random(len(u)),
u2 - sy + np.random.random(len(u)),
]
plt.plot(y)
plt.show()

dummy = np.c_[
sd + np.random.random(len(u)),
-sd + np.random.random(len(u)),
]
plt.plot(dummy)
plt.show()

以上のように,見た目では全部ぐちゃぐちゃなノイズにしか見えない3つの信号を生成した.
見た目では全部バラバラだが,に関しては"いい感じの方向"に写像できればノイズを除去できる.
それぞれだけでは"いい感じの方向"がわからないのでノイズと信号を区別できないが,両方を使って相関が大きくなる方向をいい感じの方向と定義することで,
相関する信号を抽出できる.
Scikit-Learnによる実装
sciikit-learnには既に実装されているので,以下のように簡単に正準相関と射影後の変数を求めることができる.
from sklearn.cross_decomposition import CCA
cca = CCA(n_components=1)
 間の正準相関
間の正準相関
t_x, t_y = cca.fit_transform(x,y) plt.scatter(t_x, t_y) print(np.corrcoef(t_x.T, t_y.T )[0,1]) plt.show()
正準相関の値
0.906462806701
射影した後のデータの分布

 間の正準相関
間の正準相関
t_x, t_d = cca.fit_transform(x,dummy) plt.scatter(t_x, t_d) print(np.corrcoef(t_x.T, t_y.T )[0,1]) plt.show()
正準相関の値
0.141422242479
射影した後のデータの分布

これと同じ結果を得ることを目標にpytorchで実装する.
PyTorchによる実装
pytorchにはtorch.eigという固有値を求める関数が用意されているので,これを頑張って使えば解けそうではある. が,諸々の事情からペナルティ関数法で制約条件を加えた誤差関数を設定し,autogradで微分を求めて勾配法で解く.
元の最適化問題はこれだった.
制約条件をペナルティ関数にして,以下のような誤差関数を最適化する.
は適当に0.5にした.小さい値だと制約があまり満たされず,逆に大きい値とかにするとうまく収束しないことが多い.この辺がよくわからないので,「機械学習のための連続最適化」という本を買ったけどまだ読めていない.
optimizerは何でもいいけど,単純にSGDにした.pytorch等で実装してると,なんとなくでoptimizerを選べるのでいいなと思う.
x -= np.mean(x, axis=0) y -= np.mean(y, axis=0) dummy -= np.mean(dummy, axis=0) N = len(x) def CCA(x,y, nb_steps = 100000, lr = 0.001, γ = 0.5): vxx = np.dot(x.transpose(), x)/N vyy = np.dot(y.transpose(), y)/N vxy = np.dot(x.transpose(), y)/N X = Variable(torch.from_numpy(x)).float() Y = Variable(torch.from_numpy(y)).float() a = Variable(torch.randn(2,1), requires_grad=True) b = Variable(torch.randn(2,1), requires_grad=True) Vxx = Variable(torch.from_numpy(vxx)).float() Vyy = Variable(torch.from_numpy(vyy)).float() Vxy = Variable(torch.from_numpy(vxy)).float() hist = [] from torch import optim optimizer = optim.SGD([a, b], lr = 0.0001) for i in tqdm(range(nb_steps)): Loss = - torch.mm(torch.mm(torch.t(a), Vxy), b) \ + γ * torch.abs(1 - torch.mm(torch.mm(torch.t(a),Vxx),a)) \ + γ * torch.abs(1 - torch.mm(torch.mm(torch.t(b),Vyy),b)) hist.append(Loss.data.numpy()[0,0]) optimizer.zero_grad() Loss.backward() optimizer.step() x_t = np.dot(a.data.numpy().transpose(), x.transpose()) y_t = np.dot(b.data.numpy().transpose(), y.transpose()) cca_score = torch.mm(torch.mm(torch.t(a), Vxy), b) logger.info("a Vxx a: %s"%torch.mm(torch.mm(torch.t(b),Vyy),b).data.numpy()) logger.info("b Vyy b: %s"%torch.mm(torch.mm(torch.t(a),Vxx),a).data.numpy()) return hist, x_t, y_t, cca_score
 間の正準相関
間の正準相関
hist, x_t, y_t, cca_score = CCA(x,y) plt.plot(hist) plt.show()
100%|██████████| 100000/100000 [00:19<00:00, 5232.01it/s] INFO a Vxx a: [[ 1.00017715]] INFO b Vyy b: [[ 1.00021267]]
lossの履歴

plt.scatter(x_t, y_t)
plt.show()
print(cca_score)
正準相関の値
Variable containing: 0.9066 [torch.FloatTensor of size 1x1]
射影した後のデータの分布

hist, x_t, y_t, cca_score = CCA(x,dummy) plt.plot(hist) plt.show()
100%|██████████| 100000/100000 [00:19<00:00, 5199.69it/s] INFO a Vxx a: [[ 1.00273335]] INFO b Vyy b: [[ 0.99813366]]
lossの履歴

 間の正準相関
間の正準相関
plt.scatter(x_t, y_t) plt.show() cca_score
正準相関の値
Variable containing: 1.00000e-02 * 9.6734 [torch.FloatTensor of size 1x1]
射影した後のデータの分布

無事にpytorchによる実装で,ある程度正しそうな結果を得ることができた.
おわりに
pytorchで勾配法を使ったCCAの実装をやった. 制約付き最適化を勾配法で解く良いやり方がいまいちわかってないので,勉強したい.
参考
EmacsでブラウザのテキストボックスにMarkdownを書く (校正もする)
概要
Atomic ChromeでEmacsからブラウザのテキストボタンを操作しつつ,textlintをflycheckからよんで文章を校正する. flymdでMarkdownのリアルタイムプレビューもする.
こんな感じになる.

はじめに
ブラウザのテキストボックスに文字を打ち込むのが面倒. ブラウザで操作するテキストボックスには,例えば,はてなブログやGithubのissueなどがある. これらの操作が面倒な原因として
などがある.これらの問題を解決する方法として以下をやる.
- Atomic ChromeでEmacsからchromeのテキストボックスを操作する
- textlintでEmacsから文法チェックする
- flymdでEmacsからMarkdownのリアルタイムプレビューする
(Emacs最高)
Atomic ChromeでEmacsからchromeのテキストボックスを操作する
chromeにはAtomic chromeというプラグインがあり,これを使うことでAtomからテキストボックスを操作できる. このプラグインをEmacsから使用できるパッケージが公開されている.
.emacs.d/Cask に以下を追記してインストール.
(depends-on "atomic-chrome")
また,設定ファイルのどこかに以下を書いておくと,chrome内のプラグインのボタンを押すと勝手にEmacsにバッファが開くようになる. デフォルトでは"C-x C-s"をしても保存しないようになっているが,ファイルもほしいことが多いので,これは保存のままにしておく.
(require 'atomic-chrome) (atomic-chrome-start-server) (bind-key* "C-x C-s" 'save-buffer)
Emacsで書けばキーバインドだけでなく,表をOrgTblで書いたりもできるので良い. Emacsで書いたものをはてなのテキストボックスに貼り付けてもいいけど,画像のアップロードとかが面倒なので,少しだけ楽. はてなもそうだけど,sharelatexとかもEmacsから触れるので色々良い.
textlintでEmacsから文法チェックする
textlintはMarkdownなどのテキストファイルの校正を行うアプリケーション. 本体にルールを追加していくことで,自分の用途にあった校正ができる.
作者の方が日本人であることもあって(?)日本語用のルールも充実していて, 例えば,ですますが混在しないようにするとか1文中に同じ助詞が複数でないようにするとか色々ある.
ルールを個別にインストールしていくこともできるが,上位の概念としてプリセットというのがあり,例えば日本語技術文書向けルールのプリセットなどがあり,かつその中に含まれるルールを個別にオフしたりもできるので,これを使うのが良さそう.
以下のようにインストールする.
$ npm i -g textlint textlint-rule-preset-japanese textlint-rule-preset-jtf-style textlint-rule-preset-ja-technical-writing textlint-spellcheck-tech-word textlint-rule-prh
~/.textlintrc に
{
"rules": {
"preset-japanese": {
"sentence-length": false
},
"spellcheck-tech-word": true,
"preset-ja-technical-writing": {
"ja-no-mixed-period": {
"periodMark": ".",
},
"sentence-length": false
},
"prh": {
"rulePaths" :["~/prh.yml"]
}
}
}
とか書いておくと色々動く. 普通に使うときは以下のような感じで使える.
$ textlint hoge.md
こんな感じになる.

これをEmacsから使う方法としてflycheckのルールにする方法がある. ルールの設定はEmacsからでも↑のやつを勝手によんでくれるので,以下のような感じにする.
(require 'flycheck)
(flycheck-define-checker textlint
"A linter for prose."
:command ("textlint" "--format" "unix" source-inplace)
:error-patterns
((warning line-start (file-name) ":" line ":" column ": "
(id (one-or-more (not (any " "))))
(message (one-or-more not-newline)
(zero-or-more "\n" (any " ") (one-or-more not-newline)))
line-end))
:modes (text-mode markdown-mode))
(add-to-list 'flycheck-checkers 'textlint)
(add-hook 'markdown-mode-hook 'flycheck-mode)
こんな感じで校正してくれる.

flymdでEmacsからMarkdownのリアルタイムプレビューする
もともとMarkdown書くときは使っているのでFlymdを使っている. (できればEmacs内でなんらかのcssが反映されたプレビューを見たい).
以上全部やると,最初のような感じになる.
既知の問題点
- 一旦ファイルを保存しないとtextlintが動かない.
- flymdはfirefoxで動くので,chromeとfirefox,Emacsを開いていて頭が悪い
- 特に表記ゆれについて,自分でやりがちなものを登録していくともっといい感じになると思う.
参考
- GitHub - tuvistavie/atomic-chrome: Edit Chrome textareas in Atom
- Atomic Chrome for Emacs - EmacsでChromeのテキストエリアをリアルタイム編集 - Qiita
- textlint - pluggable linting tool for text and markdown
- textlintで日本語の文章をチェックする | Web Scratch
- GitHub - textlint-ja/textlint-rule-no-mix-dearu-desumasu: textlint rule that check no mix である and ですます.
- GitHub - textlint-ja/textlint-rule-no-doubled-joshi: 文中に同じ助詞が複数出てくるのをチェックするtextlintルール
- GitHub - textlint-ja/textlint-rule-preset-ja-technical-writing: 技術文書向けのtextlintルールプリセット
- textlint for emacs flycheck · GitHub
- GitHub - mola-T/flymd: flymd - Emacs on the fly markdown preview
- EmacsでPythonとTeXとMarkdownを書いてる動画と使ってるパッケージ - やったことの説明
CycleGANで普通の木を満開の桜にする
はじめに
定期的に生成系のタスクで遊びたくなる. 今回はCycle GANを使って、普通の木を満開の桜に変換してみることにした。
Cycle GAN
論文はこれ. 中身についてはたくさん解説記事があるので、そちらを参考。
Cycle GANでは2つのドメインの間の写像を学習する。 普通のGANとは異なり(乱数ではなく)、片方のドメインの画像を入力して、もう1方のドメインの画像を出力するGeneratorがある。 また、普通のGANの誤差関数(discriminatorの判別)だけでなく、1度片方のGeneratorに通した後、もう一方のGeneratorに通して再構成誤差を計算し、誤差関数に加える。
pix2pixなどでは対になる画像を用意しないと学習ができないが、CycleGANではそういうのがいらないという利点がある。
実験
ImageNetから桜の画像3000枚と普通の木の画像2500枚をダウンロードした. 画像をざっと見た感じ,桜は木全体だけでなく花だけアップの写真が多くて少し気になった. また,桜と富士山が写っている画像も結構多い.
著者の実装がgithubで公開されているので,それにダウンロードしたデータを食わせる. gtx780tiで2日ぐらい学習させた. 古いGPUでGPUメモリが全然足りないので,オプションで色々設定した。(そろそろ買いたい)
$ python train.py --dataroot ./datasets/tree2sakura --name tree2sakura --model cycle_gan --no_dropout --pool_size 50 --loadSize 128 --fineSize 128
結果
1エポック後
| 元画像 | 生成画像 | 再構成画像 |
|---|---|---|
 |
 |
 |
 |
 |
 |
50エポック後
| 元画像 | 生成画像 | 再構成画像 |
|---|---|---|
 |
 |
 |
 |
 |
 |
100エポック後
| 元画像 | 生成画像 | 再構成画像 |
|---|---|---|
 |
 |
 |
 |
 |
 |
100エポックのネットワークで普通の木を桜に変換したもの
白と黒を変換しがちなのが少し気になるけど,なかなかうまくできたと思う.
良さそうなもの
| 元画像 | 生成画像 |
|---|---|
 |
 |
 |
 |
よくなさそうなもの
| 元画像 | 生成画像 |
|---|---|
 |
 |
 |
 |
空が白っぽいとその部分を黒で塗りつぶす傾向がある。なぜかはわからない。
おわりに
既存のコードを動かしただけだけど、思ったよりもうまく動いた。 が、空が異常に暗くなるなど、できてないやつも多い。体感では半々ぐらい。
せっかく対応する画像がなくても学習できるので、こちらでやられている犬猫変換のように、それなりに離れたドメイン間でやると面白そう。
生成系の研究はレッドオーシャンな感じで自分でやるのは大変そうだけど, 公開されているもので遊ぶのは楽しいので、ちょいちょいやっていきたい.
参考
pythonでNUTSの実装
はじめに
今回は No U-Turn Sampler (NUTS)の実装をやる. 論文を参考にした.
コードはここにもある
NUTS
ハミルトニアンモンテカルロ (HMC)はパラメータの勾配を利用して, 効率的にMCMCサンプリングを行うことができる手法だった.
HMCの問題点は2つ.
- 更新ステップ数
を適切に指定しなければいけない.
- 更新の大きさ
を適切に指定しなければいけない.
NUTSは更新ステップ数を自動的に決定する手法である.
論文内では
はdual-averaging (Nesterov 2009)を用いて決定するが,今回は決め打ちにする.
更新ステップ数L
ハミルトニアンモンテカルロでは,正規分布によって発生させた運動量を与えて,
ステップの間,点を動かす.
予め決められた
ステップの間,点を動かすので,例えば谷にハマった時などガタガタして無駄な計算をしてしまう.
NUTSでは「Uターンしたら点を動かすのをやめる」という規則でこの無駄な計算をなくす. ただし,単純にUターンしたときに中断したら詳細釣り合い条件を満たさなくなるので,少し工夫する.
具体的には,だいたい
- 運動量
をランダムに決める.
- 時間の向き
を{-1, 1}からランダムに選ぶ.
- 選んだ時間の向きの方向に
回,点を移動させる(点の軌跡は全て記憶しておく)
- どこかでUターンしていないかを確認する
- Uターンしていたら6へ,それ以外は
して2へ戻る
- これまでに計算した軌跡からランダムに1点選んで,サンプリング結果に加えて,1に戻る
という感じ.
実装
以下のようなデータの平均と分散をサンプリングする.
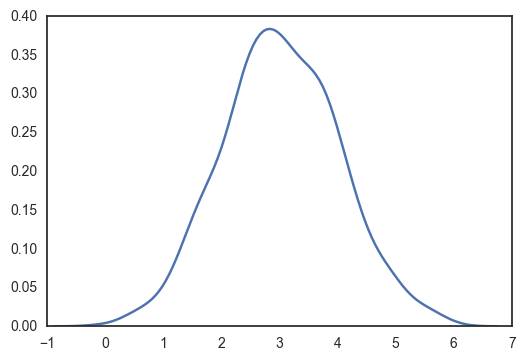
import numpy as np from numpy import exp from copy import deepcopy import pylab as plt import seaborn as sns from scipy.stats import norm, gamma from tqdm import tqdm from numpy import random from matplotlib.animation import FuncAnimation sns.set_style("white") true_μ = 3 true_σ = 1 nb_data = 1000 x = np.random.normal(true_μ, true_σ, nb_data) print(x.mean(), x.std()) sns.kdeplot(x) plt.show()
2.99882646635 1.02180321215

NUTSのプログラムは以下. 論文のNaive-NUTSの実装をする. Leapfrogで点を移動させるなどはハミルトニアンモンテカルロと同じ.
def log_dh(μ, σ): return np.array([-np.sum(x - μ) / σ**2, len(x) / (2 * σ**2) - np.sum((x - μ)**2) / (2 * σ**4)]) def H(θₜ, p): return -norm_lpdf(θₜ[0], θₜ[1]) + 0.5 * np.dot(p, p) def Leapfrog(x, θ, r, ε): θ_d = deepcopy(θ) r_d = deepcopy(r) r_d -= 0.5 * ε * log_dh(θ_d[0], θ_d[1]) θ_d[0] = θ_d[0] + ε * r_d[0] θ_d[1] = θ_d[1] + ε * r_d[1] r_d -= 0.5 * ε * log_dh(θ_d[0], θ_d[1]) return θ_d, r_d
norm_lpdf = lambda μ, σ: np.sum(norm.logpdf(x, μ, σ)) gamma_lpdf = lambda a: np.sum(gamma.logpdf(x, a)) Δ_max = 1000 ε = 0.05 L = norm_lpdf M = 100 θ0 = np.array([random.randn(), random.gamma(1)]) list_θₘ = [θ0]
回の移動は再帰で実装される.
def BuildTree(θ, r, u, v, j, ε): if j == 0: θd, rd = Leapfrog(x, θ, r, v * ε) if np.log(u) <= (L(*θd) - 0.5 * np.dot(rd, rd)): Cd_ = [[θd, rd]] else: Cd_ = [] sd = int(np.log(u) < (Δ_max + L(*θd) - 0.5 * np.dot(rd, rd))) return θd, rd, θd, rd, Cd_, sd else: θ_minus, r_minus, θ_plus, r_plus, Cd_, sd = BuildTree(θ, r, u, v, j - 1, ε) if v == -1: θ_minus, r_minus, _, _, Cdd_, sdd = BuildTree(θ_minus, r_minus, u, v, j - 1, ε) else: _, _, θ_plus, r_plus, Cdd_, sdd = BuildTree(θ_plus, r_plus, u, v, j - 1, ε) sd = sdd * sd * int((np.dot(θ_plus - θ_minus, r_minus) >= 0) and (np.dot(θ_plus - θ_minus, r_plus) >= 0)) Cd_.extend(Cdd_) return θ_minus, r_minus, θ_plus, r_plus, Cd_, sd
hist_L = [] hist_C = [] for m in tqdm(range(M)): r0 = random.randn(2) u = random.uniform(0, exp(L(*list_θₘ[-1]) - 0.5 * np.dot(r0, r0))) θ_minus = deepcopy(list_θₘ[-1]) θ_plus = deepcopy(list_θₘ[-1]) r_minus = deepcopy(r0) r_plus = deepcopy(r0) j = 0 C = [[deepcopy(list_θₘ[-1]), deepcopy(r0)]] s = 1 while s == 1: v = random.choice([-1, 1]) if v == -1: θ_minus, r_minus, _, _, Cd, sd = BuildTree(θ_minus, r_minus, u, v, j, ε) else: _, _, θ_plus, r_plus, Cd, sd = BuildTree(θ_plus, r_plus, u, v, j, ε) if sd == 1: C.extend(Cd) s = sd * int((np.dot(θ_plus - θ_minus, r_minus) >= 0) and (np.dot(θ_plus - θ_minus, r_plus) >= 0)) j += 1 index = random.choice(list(range(len(C)))) list_θₘ.append(C[index][0]) hist_L.append(L(C[index][0][0], C[index][0][1])) hist_C.append(C)
100%|██████████| 100/100 [00:00<00:00, 760.56it/s]
def plot(list_θₘ, hist_C): fig = plt.figure() ax = fig.add_subplot(111) def update(i): fig.canvas.draw() ax.cla() j = i // 3 if (i % 3) == 0: ax.scatter(np.array(hist_C[j])[:, 0, 0], np.array(hist_C[j])[:, 0, 1], linewidth=0, marker=".") ax.plot(list_θₘ[:(j), 0], list_θₘ[:(j), 1], c="gray", linewidth=0.3, alpha=0.4) ax.scatter(list_θₘ[:(j), 0], list_θₘ[:(j), 1], c="g", linewidth=0, marker=".") elif (i % 3) == 1: ax.scatter(np.array(hist_C[j])[:, 0, 0], np.array(hist_C[j])[:, 0, 1], linewidth=0, marker=".") ax.plot(list_θₘ[:(j + 1), 0], list_θₘ[:(j + 1), 1], c="gray", linewidth=0.3, alpha=0.4) ax.scatter(list_θₘ[:(j + 1), 0], list_θₘ[:(j + 1), 1], c="g", linewidth=0, marker=".") else: ax.scatter(np.array(hist_C[j])[:, 0, 0], np.array(hist_C[j])[:, 0, 1], linewidth=0, marker=".", c="w") ax.plot(list_θₘ[:(j + 1), 0], list_θₘ[:(j + 1), 1], c="gray", linewidth=0.3, alpha=0.4) ax.scatter(list_θₘ[:(j + 1), 0], list_θₘ[:(j + 1), 1], c="g", linewidth=0, marker=".") plot_lim = 30 if(j) > plot_lim: temp_list = np.array([[np.min(np.array(C)[:, 0], axis=0), np.max(np.array(C)[:, 0], axis=0)] for C in hist_C[j - plot_lim: j]]) temp_xlim = [np.min(temp_list[:, :, 0]), np.max(temp_list[:, :, 0])] xlim_range = temp_xlim[1] - temp_xlim[0] temp_ylim = [np.min(temp_list[:, :, 1]), np.max(temp_list[:, :, 1])] ylim_range = temp_ylim[1] - temp_ylim[0] plt.xlim(temp_xlim[0] - xlim_range * 0.1, temp_xlim[1] + xlim_range * 0.1) plt.ylim(temp_ylim[0] - ylim_range * 0.1, temp_ylim[1] + ylim_range * 0.1) ani = FuncAnimation(fig, update, frames=len(hist_C) * 3 - 2) ani.save("temp.gif", writer="imagemagick", fps=3) plot(list_θₘ, hist_C)
NUTSのプロット。なんかU-Turnしているけど、アルゴリズムをよく見ると、一番上の木でU-Turnしても棄却しないようになってるし、あってるはず pic.twitter.com/p885RrqrVf
— けいの (@ksknw) 2017年8月5日
たぶんできていると思う きれいに書き直すつもりだったけど、面倒だったからやめた
「2017年3月14日をもって動画のアップロード機能は終了いたしました」 じゃないんだよなぁ
参考
ハミルトニアンモンテカルロ法の実装
はじめに
今までなんとなくStanなどを使ってMCMCをやっていた。 ギブスサンプリングぐらいなら昔勉強したけど、ハミルトニアンモンテカルロや、ましてやNUTSなどは何をやっているのかあまり理解していなかった。 基礎からのベイズ統計学という本を読んで、ハミルトニアンモンテカルロまではなんとなくわかったので、プログラムを書いて理解する。
NUTSはまだわかってないので、
を実装する。
理論は他の方のブログや本を参考にしてほしい。 自分の理解で適当に書いているので、間違ってたらごめんなさい。
データ
なんでもいいけど、平均3標準偏差1の1次元の正規分布に従うデータから、その平均と分散を推定することにする。
import numpy as np from numpy import random from copy import deepcopy import pylab as plt import seaborn as sns from matplotlib.animation import FuncAnimation from scipy.stats import norm, gamma from tqdm import tqdm sns.set_style("white") true_μ = 3 true_σ = 1 nb_data = 1000 data = np.random.normal(true_μ, true_σ, nb_data) print(data.mean(), data.std()) sns.kdeplot(data) plt.show()
3.02683958884 0.998470831061
/home/kei/anaconda3/lib/python3.5/site-packages/statsmodels/nonparametric/kdetools.py:20: VisibleDeprecationWarning: using a non-integer number instead of an integer will result in an error in the future
y = X[:m/2+1] + np.r_[0,X[m/2+1:],0]*1j
メトロポリスヘイスティングス法
からパラメータの候補aを生成する
を計算する。
- 確率
でaを受容し、
とする。
を繰り返してパラメータを更新していく。
よって、現在のパラメータから新しいパラメータを生成するときの確率、および、尤度
を計算するときの分布の形状(提案分布)を決めると、パラメータを推定することができる。
今回は提案分布を正規分布として、その平均と標準偏差を推定する。
独立メトロポリスヘイスティングス法
まずは、つまり次のパラメータを前のパラメータに依存せず独立にサンプリングする。
今回は平均と標準偏差の推定なので、それぞれ正規分布およびガンマ分布からパラメータをサンプリングする。
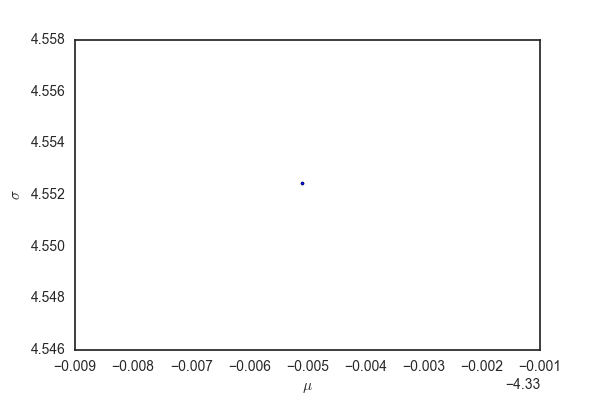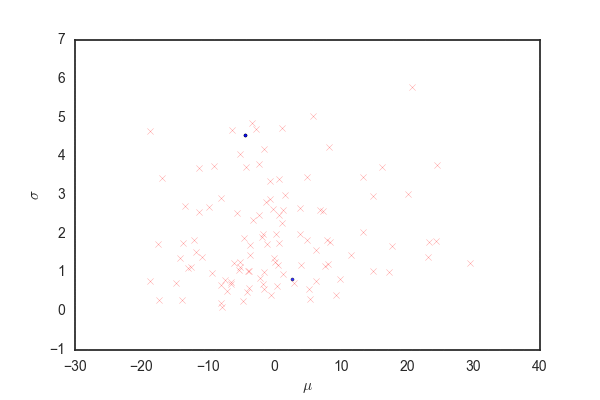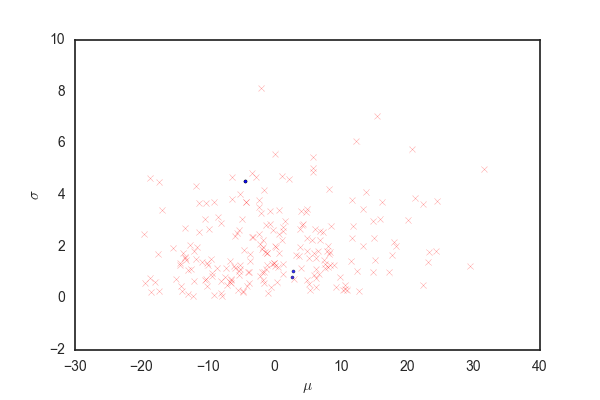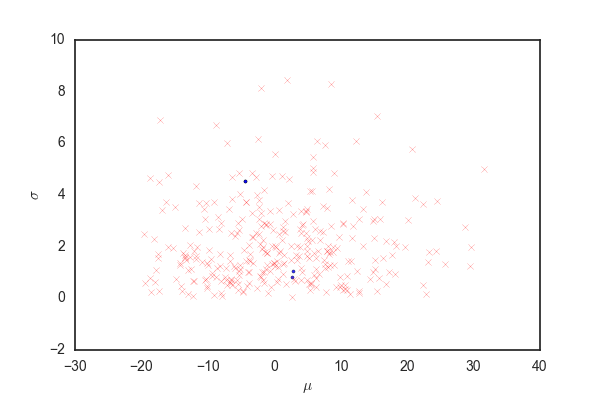
sampling_μ_mean = 0 sampling_μ_std = 10 sampling_σ_a = 2 norm_lpdf = lambda x, μ, σ: np.sum(norm.logpdf(x, μ, σ)) gamma_lpdf = lambda x, a: np.sum(gamma.logpdf(x, a)) logf = lambda θₜ: norm_lpdf(data, θₜ["μ"], θₜ["σ"]) logq = lambda θₜ: norm_lpdf(θₜ["μ"], sampling_μ_mean, sampling_μ_std) \ + gamma_lpdf(θₜ["σ"], sampling_σ_a) logr = lambda a, θₜ: logq(θₜ) + logf(a) - logq(a) - logf(θₜ) nb_samples = 1000 θₜ = {} a = {} # 候補点 hist = [] θₜ["μ"] = random.normal(sampling_μ_mean, sampling_μ_std) θₜ["σ"] = random.gamma(sampling_σ_a) for t in tqdm(range(nb_samples)): a["μ"] = random.normal(sampling_μ_mean, sampling_μ_std) a["σ"] = random.gamma(sampling_σ_a) if logr(a, θₜ) > 0: θₜ = deepcopy(a) hist.append([a["μ"], a["σ"], 0]) else: if np.exp(logr(a, θₜ)) > random.uniform(0, 1): θₜ = deepcopy(a) hist.append([a["μ"], a["σ"], 1]) else: hist.append([a["μ"], a["σ"], 2]) hist = np.array(hist) print(θₜ)
100%|██████████| 1000/1000 [00:03<00:00, 323.83it/s]
{'σ': 1.1803787969064548, 'μ': 2.801370701839039}
サンプリングがどのように進んだかをプロットしてみる。
def update(i, hist): if hist[i, 2] == 0: plt.scatter(hist[i, 0], hist[i, 1], marker=".", c="b", alpha=0.8) elif hist[i, 2] == 1: plt.scatter(hist[i, 0], hist[i, 1], marker=".", c="g", alpha=0.8) elif hist[i, 2] == 2: plt.scatter(hist[i, 0], hist[i, 1], marker="x", c="r", alpha=0.4) fig = plt.figure() plt.xlabel(r"$\mu$") plt.ylabel(r"$\sigma$") ani = FuncAnimation(fig, update, fargs=(hist,), frames=nb_samples) ani.save("independent.gif", writer="imagemagick")
青い点が受容されたパラメータ、赤いバツが棄却された候補を表している。 殆どの候補が棄却されて、むちゃくちゃ効率が悪いことがわかる。
ランダムウォークMH法
として、新しいパラメータ候補
を生成すると、これまで見つかった良さそうなパラメータの周辺を探索することになるので、受容率が上がって効率のいいサンプリングが行える。
今回はが平均0、標準偏差0.05の正規分布に従うとしてサンプリングを行った。
このとき、
になる。
も同様。
sampling_mean = 0 sampling_std = 0.05 logf = lambda θₜ: norm_lpdf(data, θₜ["μ"], θₜ["σ"]) logq = lambda a, θₜ: norm_lpdf(θₜ["μ"], sampling_mean+a["μ"], sampling_std) \ + norm_lpdf(θₜ["σ"], sampling_mean+a["σ"], sampling_std) logr = lambda a, θₜ: logq(θₜ,a) + logf(a) - logq(θₜ,a) - logf(θₜ) nb_samples = 1000 θₜ = {} a = {} # 候補点 hist = [] hist_2sigma = [] nb_i = 40 sample_2sigma = [np.array([2*sampling_std*np.sin(i*2*np.pi/nb_i), 2*sampling_std*np.cos(i*2*np.pi/nb_i)]) for i in range(nb_i)] θₜ["μ"] = random.normal(1, 1) θₜ["σ"] = random.gamma(3) for t in tqdm(range(nb_samples)): a["μ"] = θₜ["μ"] + random.normal(sampling_mean, sampling_std) a["σ"] = θₜ["σ"] + random.normal(sampling_mean, sampling_std) if logr(a, θₜ) > 0: θₜ = deepcopy(a) hist.append([a["μ"], a["σ"], 0]) else: if np.exp(logr(a, θₜ)) > random.uniform(0, 1): θₜ = deepcopy(a) hist.append([a["μ"], a["σ"], 1]) else: hist.append([a["μ"], a["σ"], 2]) hist_2sigma.append([np.array([θₜ["μ"],θₜ["σ"]]) + sample_2sigma_i for sample_2sigma_i in sample_2sigma]) hist = np.array(hist) hist_2sigma = np.array(hist_2sigma) print(θₜ)
100%|██████████| 1000/1000 [00:01<00:00, 592.47it/s]
{'σ': 0.9944398277566455, 'μ': 3.0750991483862506}
def plot(hist, hist_2sigmam, filename, T=1): lim_range = 0.2 fig = plt.figure() ax = fig.add_subplot(111) fill, = ax.fill(hist_2sigma[0, :, 0], hist_2sigma[0, :, 1], alpha=0.2, c="gray") plt.xlabel(r"$\mu$") plt.ylabel(r"$\sigma$") def update(i, hist, hist_2sigma, line): fill.set_xy(hist_2sigma[i]) fig.canvas.draw() if hist[i, 2] == 0: ax.scatter(hist[i, 0], hist[i, 1], marker=".", c="b", alpha=0.8, linewidths=0) elif hist[i, 2] == 1: ax.scatter(hist[i, 0], hist[i, 1], marker=".", c="g", alpha=0.8, linewidths=0) else: # hist[i, 2] == 2: ax.scatter(hist[i, 0], hist[i, 1], marker="x", c="r", alpha=0.4) plot_lim = 10 if i > plot_lim: plt.xlim(min(min(hist[i - plot_lim:i, 0]), 3-lim_range), max(max(hist[i - plot_lim:i, 0]), 3+lim_range)) plt.ylim(min(min(hist[i - plot_lim:i, 1]), 1-lim_range), max(max(hist[i - plot_lim:i, 1]), 1+lim_range)) ani = FuncAnimation(fig, update, fargs=(hist, hist_2sigma, line), frames=nb_samples//T) ani.save(filename, writer="imagemagick")
plot(hist, hist_2sigma, "random_walk.gif", 3)
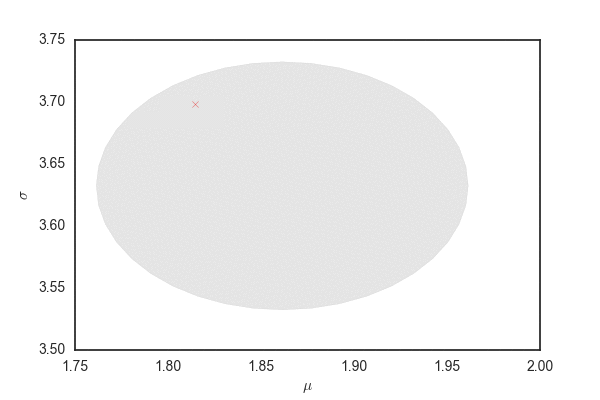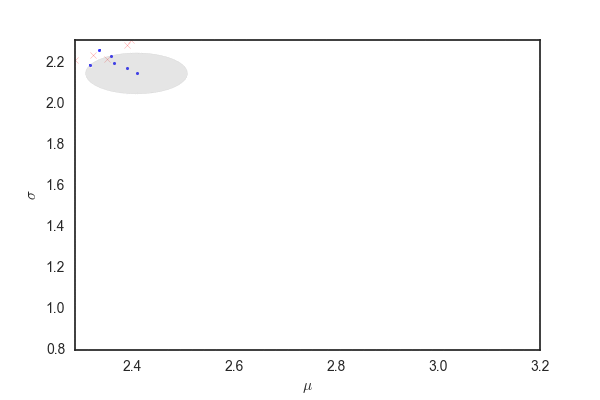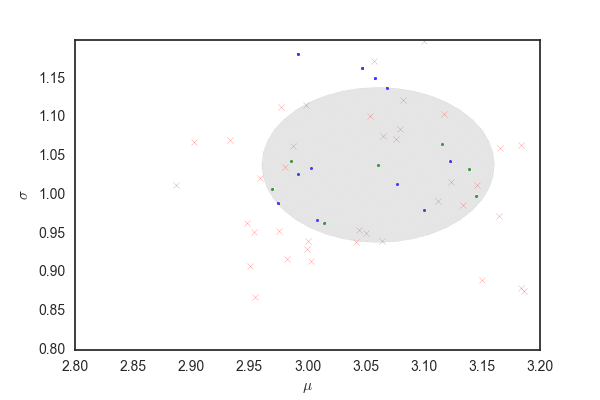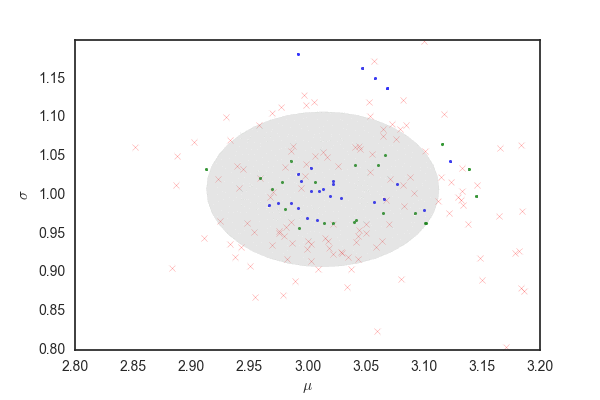
青い点および赤いバツは上と同じ、確率rで受容されたパラメータ。 96%ぐらいのパラメータの候補が灰色の領域から選ばれる。 先ほどと比べるとだいぶ多くのパラメータが受容されている。
プロットの範囲がいまいちなのは、どうすればいい感じになるのかよくわからなかったからです…
ハミルトニアンモンテカルロ法
ランダムウォークMH法はを適切に設定しないと特にパラメータの次元が高くなるとうまくいかない(らしい)。
パラメータによる勾配が利用できるのであれば、それを使ってパラメータを更新すると良さそうである。
ハミルトニアンモンテカルロ法では慣性付きの勾配法っぽい感じでパラメータを更新する。
- モーメンタム
を標準正規乱数から発生させる。
- ポテンシャル
を対数事後分布として、慣性付きの勾配法を進めて、
ステップ後の値を
とする。
を求める。
- 確率
でパラメータ
を受容する。
ここではハミルトニアンで
普通の勾配法と違うのは、
ステップ更新するごとに、慣性項を標準正規乱数でリセットする。
- まれに更新を棄却する
- 粘性項がない
といったところかと思う。
(普通の確率勾配法で更新したらいかんのかと思ってしまうけど、そういう研究もあるんだろうな)
def log_dh(x, μ, σ): return np.array([-np.sum(x - μ) / σ**2, len(x) / (2 * σ**2) - np.sum((x - μ)**2) / (2 * σ**4)]) def H(x, θₜ, p): return -norm_lpdf(x, θₜ["μ"], θₜ["σ"]) + 0.5 * np.dot(p, p) nb_samples = 1000 θₜ = {} θₐ = {} ε = 0.01 L = 100 hist = [] hist_2sigma = [] θₜ["μ"] = random.normal(1, 1) θₜ["σ"] = random.gamma(3) nb_i = 40 p_2sigma = [np.array([2*np.sin(i*2*np.pi/nb_i), 2*np.cos(i*2*np.pi/nb_i)]) for i in range(nb_i)] def update(pₜ, θₜ): pₐ = deepcopy(pₜ) θₐ = deepcopy(θₜ) for t in range(L): pₐ -= ε / 2 * log_dh(data, θₐ["μ"], θₐ["σ"]) dμ, dσ = ε * pₐ θₐ["μ"] += dμ θₐ["σ"] += dσ θₐ["σ"] = np.abs(θₐ["σ"]) pₐ -= ε / 2 * log_dh(data, θₐ["μ"], θₐ["σ"]) return pₐ, θₐ for i in tqdm(range(nb_samples)): pₜ = np.random.randn(2) pₐ, θₐ = update(pₜ, θₜ) if np.exp(H(data, θₜ, pₜ) - H(data, θₐ, pₐ))>1: θₜ = deepcopy(θₐ) hist.append([θₐ["μ"], θₐ["σ"], 0]) elif np.exp(H(data, θₜ, pₜ) - H(data, θₐ, pₐ)) > random.uniform(0,1): θₜ = deepcopy(θₐ) hist.append([θₐ["μ"], θₐ["σ"], 1]) else: hist.append([θₐ["μ"], θₐ["σ"], 2]) list_θ = [update(p_2sigma_i, θₜ)[1] for p_2sigma_i in p_2sigma] hist_2sigma.append(np.array([[θ["μ"],θ["σ"]] for θ in list_θ])) hist = np.array(hist) hist_2sigma = np.array(hist_2sigma) print(θₜ)
100%|██████████| 1000/1000 [05:34<00:00, 2.24it/s]
{'σ': 1.0048874933483771, 'μ': 3.0559467768730531}
plot(hist, hist_2sigma, "hamiltonian.gif", 3)
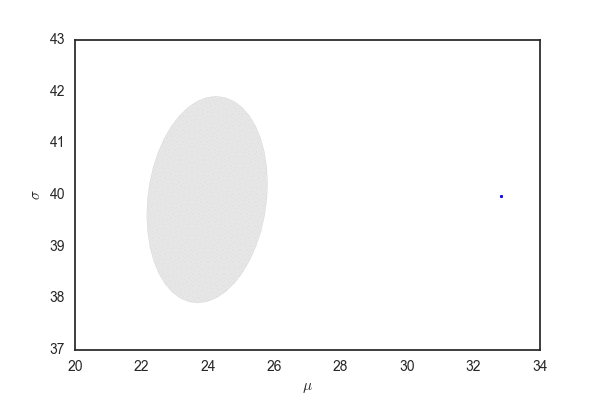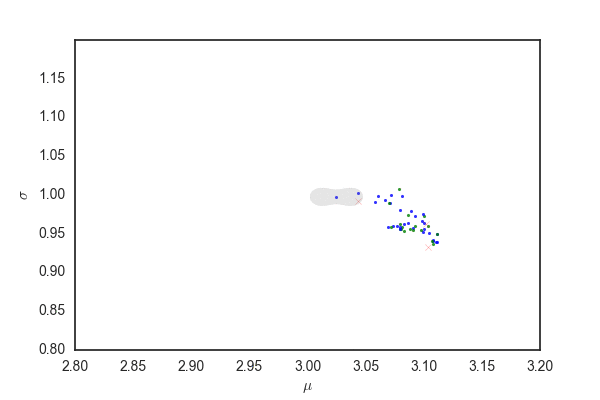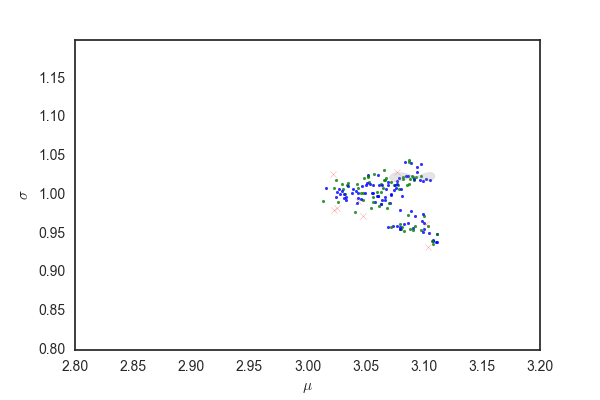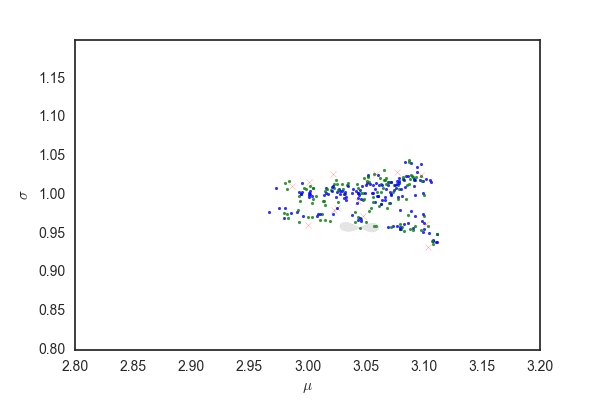
だいたい今までと同じだけど、灰色の部分はpを発生させるときに96%ぐらいにしているので、勾配法で更新されたあとでこの範囲にサンプリングが収まっているかは保証されないと思う。 matplotlibのfill関数の使い方が間違っていなければ、最初のほうでサンプリングの範囲がぐちゃぐちゃになっている部分があるが、これは関数がぐちゃぐちゃだから(??)
ランダムウォークと比べても、棄却されるサンプルが圧倒的に少なく効率的にサンプリングできている。
おわりに
自分の勉強のために
を実装した。
勾配法との関係がいまいちわかっていなくて、例えばAdamとか使っても詳細釣り合い条件が満たされるのかよくわからないし、そもそも勾配法だけではいけないのか。 次はNUTSを理解したい。
参考
pytorchでgmmの最尤推定
はじめに
今まではKerasを使っていたけど、最近になってpytorchを覚えようとしている。 “Define by Run"と"Define and Run"の違いとかはよくわかっていないのでそのへんは適当。
普通にtutorialだけやっていると、 “なんとかネットワークは作れるけど、自分が考えた新しい層を追加できない” ということになりそうだったので、ネットにあまり情報のなかったgmmを勾配法(最尤推定)で解くプログラムを作って、pytorchを理解することにした。
gaussian mixture model
適当にデータを作る
%matplotlib inline import pylab as plt import seaborn as sns sns.set_style("white") from scipy.stats import norm import numpy as np import torch from torch.autograd import Variable K = 2 nb_data = 2000 nb_steps = 2000 true_μ_K = [-3, 3] true_σ_K = [1, 1] true_π_K = [0.5, 0.5] np_data = [] for μₖ, σₖ, πₖ in zip(true_μ_K, true_σ_K, true_π_K): for i in range(int(nb_data * πₖ)): np_data.append(np.random.normal(μₖ, σₖ)) np_data = np.array(np_data)
def gmm_plot(list_mean, list_std, list_pi, **kwargs): x = np.linspace(-10,10,500) y = np.sum([norm.pdf(x, mean, np.abs(std))*pi for mean,std,pi in zip(list_mean, list_std, list_pi)], axis=0) return plt.plot(x,y, **kwargs) gmm_plot(true_μ_K, true_σ_K, true_π_K)

最尤推定
勾配法でパラメータを推定するための誤差関数として、今回は単純な尤度を使った。
gmmの尤度は
。これと、πの合計が1になるように、適当に制約を加えて、以下のように誤差関数を定義した。
def get_normal_lpdf(x_N, μ, σ): μ_N = μ.expand(x_N.size()) σ_N = σ.expand(x_N.size()) return -0.5 * torch.log(2 * np.pi * σ_N ** 2) - 0.5 * (x_N - μ_N)**2 / σ_N ** 2 def get_loss(normal_lpdf_K_N, π_K): gmm_lpdf_N = 0 for normal_lpdfₖ_N, πₖ in zip(normal_lpdf_K_N, π_K): πₖ_N = πₖ.expand(normal_lpdfₖ_N.size()) gmm_lpdf_N += (torch.exp(normal_lpdfₖ_N) * πₖ_N) # TODO logsumexpを実装したほうがいいかも gmm_lpdf = torch.mean(torch.log(gmm_lpdf_N)) Σπ = torch.sum(π_K) gmm_lpdf -= torch.abs(1 - Σπ) # 制約条件 return -gmm_lpdf
pytorchではautograd.Variableで変数を定義しておくと、勝手に微分を計算してくれるらしい。 ので、以下のようにデータと求めたいパラメータを定義する。
x_N = Variable(torch.from_numpy(np_data), requires_grad=False).float()
lr = 0.05 μ_K = Variable(torch.randn(K), requires_grad=True) σ_K = Variable(torch.randn(K)**2, requires_grad=True) π_K = Variable(torch.abs(torch.randn(K)), requires_grad=True)
あとは以下のように誤差を伝搬させて、パラメータを更新する。 grad.zero_をしないといけないと知らなくて苦労した。
history_loss = [] history_μ_K = [] history_σ_K = [] history_π_K = [] for i in range(nb_steps): normal_lpdf_K_N = [] for k in range(K): normal_lpdf_K_N.append(get_normal_lpdf(x_N, μ_K[k], σ_K[k])) loss = get_loss(normal_lpdf_K_N, π_K) loss.backward() μ_K.data -= μ_K.grad.data * lr μ_K.grad.data.zero_() σ_K.data -= σ_K.grad.data * lr σ_K.grad.data.zero_() π_K.data -= π_K.grad.data * lr π_K.grad.data.zero_() π_K.data = torch.abs(π_K.data) history_loss.extend(loss.data.tolist()) history_μ_K.append(μ_K.data.tolist()) history_σ_K.append(σ_K.data.tolist()) history_π_K.append(π_K.data.tolist())
うまく収束してくれた。
gmm_plot(μ_K.data, σ_K.data, π_K.data) gmm_plot(true_μ_K, true_σ_K, true_π_K) plt.show()

plt.plot(history_loss) plt.show()

収束のアニメーションをかいてみる
import matplotlib.animation as animation plts = [] fig = plt.figure() for μ_K, σ_K, π_K in zip(history_μ_K[::10], history_σ_K[::10], history_π_K[::10]): plts.append(gmm_plot(μ_K, σ_K, π_K, c="b")) ani = animation.ArtistAnimation(fig, plts, interval=100) ani.save('anim.gif', writer="imagemagick") ani.save('anim.mp4', writer="ffmpeg") plt.show()
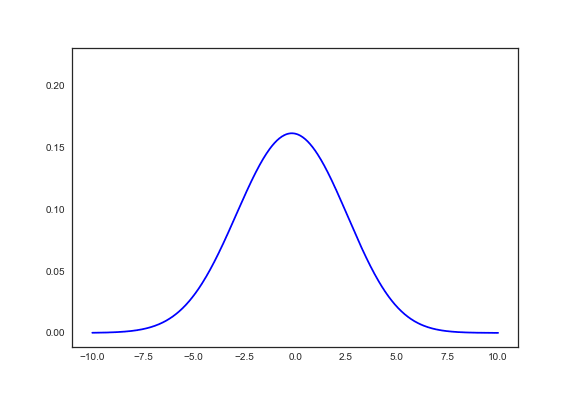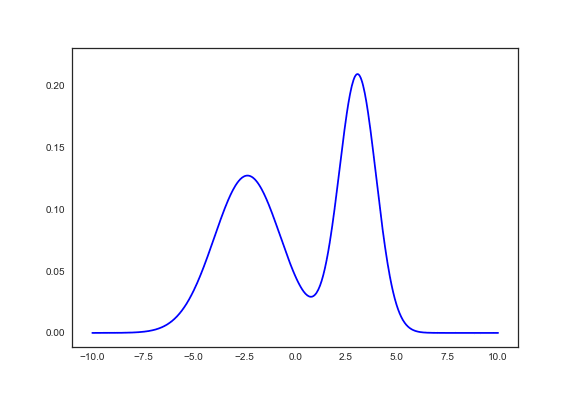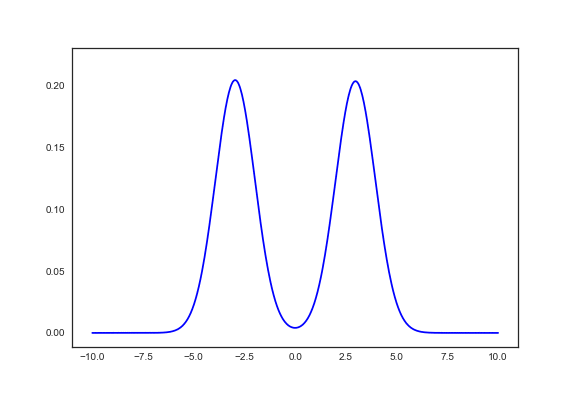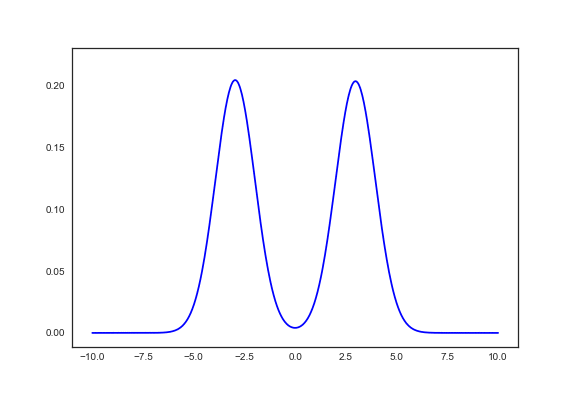
これはうまく収束した結果だけど、何度か実行すると以下のような微妙な結果に収束することも多かった。 (収束したあとに"脈打ってる"のはなんだ…)

まとめ
pytorchの使い方を覚えるために、gmmをやった。 あまり安定していないけど、動くものができた。 なんとなく使い方はわかってきたけど、"勝手に中身が書き換えられている"という印象をもってしまう部分がある。
やる気があればMAP推定、SGDもしくはHMCに続くかも。