関係データ学習の実装 PythonでStochastic Block Modelの実装
概要
関係データ学習を見ながらpythonでSBMの実装をした。
Twitterのフォローフォロワー関係データに適用して、それっぽい結果を得た。
はてなの数式がいまいちわからないので、外部でレンダリングをしていて表示が遅い。
はじめに
以前StanでStochastic Block Modelをやろうとして失敗した. すっかり忘れていたけど,ふと思い出したので,Stanではなく,普通にpythonで実装することにした. 更新式の導出などは前回と同様に「関係データ学習」を参考にした.
データ
前回と同じツイッターのフォローフォロワー関係のデータを使って、アルゴリズムをテストする。 使うのは以下のような、非対称な関係データ。
import pandas as pd data = pd.read_csv("./combinationTable.csv") uname = data[:1].get_values()[0] data.drop(0).head()
| 11213962 | 1603589724 | 68746721 | 267765193 | 10985942 | 14009672 | 167346791 | 2896013873 | 17364190 | 31442147 | ... | 118320586 | 218493756 | 232520574 | 385409365 | 419425806 | 102227818 | 301210136 | 175163526 | 252996913 | 152543735 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | False | False | True | False | False | False | False | True | False | True | ... | False | False | False | False | False | False | False | False | False | False |
| 2 | False | False | False | False | False | False | False | False | False | True | ... | False | False | False | False | False | False | False | False | False | False |
| 3 | False | False | False | False | False | True | False | False | False | False | ... | False | False | False | False | False | False | False | False | False | False |
| 4 | False | False | False | False | False | False | False | False | False | True | ... | False | False | False | False | False | False | False | False | False | False |
| 5 | False | False | True | False | False | True | True | False | False | False | ... | False | False | False | False | False | False | False | False | False | False |
5 rows × 120 columns
%matplotlib inline import pylab as plt import seaborn import numpy as np X = (data.get_values()[1:]=="True") def plot_matrix(matrix, z1=None, z2=None): f, ax = plt.subplots(figsize=(10, 10)) plt.pcolor(matrix, cmap=plt.cm.Blues ) if not z1 is None: z1_diff = np.r_[[0], np.diff(z1)] z2_diff = np.r_[[0], np.diff(z2)] for i,c in enumerate(z1_diff): if c!=0: ax.axhline(i, c="grey")#, linewidth=1) for i,c in enumerate(z2_diff): if c!=0: ax.axvline(i, c="grey") #, linewidth=1) plt.show() plot_matrix(X)

SBMの事後確率
本の内容を参考に、周辺化ギブスサンプラーによって、クラスタの割り当て,
をサンプリングする。
他の変数がgivenだとした時の、の事後確率は、
ここで、
はガンマ関数で、
はパラメータ
については対称なので省略。
以上をそのままpythonのプログラムにした。 z1とz2はまとめられそうだけど(というかXを転置して同じ関数に突っ込むだけでいいはずだけど)、ややこしくなるのが嫌だったので2つバラバラの関数として実装した。 プログラムは以下。
サンプリングの回数や独立なサンプルを得るためにサンプルを何回おきに保存するかなど、よくわからなかったので、適当に決めた。
実行結果
core-i5-7200U(2.5GHz)でだいたい5時間半かかった。 一切並列に計算していないので、むちゃくちゃ重い。 ラベルがスイッチしないようにするのが難しいかもしれないが、burn-inが終わった後から、chainを生成して並列にサンプリングしたりしてもいいのかもしれない。
import pickle with open("./sample_z.pkl", "rb") as f: samples_z1, samples_z2 = pickle.load(f) nb_k = 8
def onehot(i, nb_k): ret = np.zeros(nb_k) ret[i] = 1 return ret z1 = np.array([[onehot(i, nb_k) for i in sample] for sample in samples_z1]) z2 = np.array([[onehot(i, nb_k) for i in sample] for sample in samples_z2])
ちゃんと収束しているかを確認するためにいくつかのヒストグラムを書く。
%matplotlib inline import pylab as plt N=6 i=0 plt.hist(np.array(samples_z1)[:,N*i:N*(i+1)], linewidth=0)
([array([ 0., 0., 0., 0., 0., 38., 0., 0., 953., 0.]), array([ 555., 430., 0., 0., 0., 6., 0., 0., 0., 0.]), array([ 0., 988., 0., 0., 0., 3., 0., 0., 0., 0.]), array([ 971., 17., 0., 0., 0., 2., 0., 0., 0., 1.]), array([ 0., 988., 3., 0., 0., 0., 0., 0., 0., 0.]), array([ 0., 964., 27., 0., 0., 0., 0., 0., 0., 0.])], array([ 0. , 0.7, 1.4, 2.1, 2.8, 3.5, 4.2, 4.9, 5.6, 6.3, 7. ]), <a list of 6 Lists of Patches objects>)
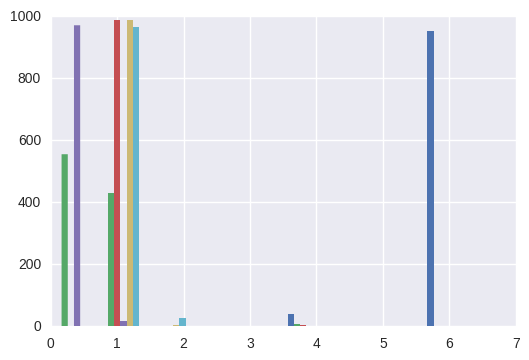
怪しい部分(緑)もある。収束していないのかもしれない。 どのようにクラスタができたかをプロットする。
z1 = z1.mean(axis=0) z2 = z2.mean(axis=0)
def sort_by_cluster(matrix, z1, z2): sorted_mat = list(zip(z1, matrix)) sorted_mat.sort(key=lambda x:x[0]) sorted_z1,sorted_mat = zip(*sorted_mat) sorted_mat = list(zip(z2, np.array(sorted_mat).T)) sorted_mat.sort(key=lambda x:x[0]) sorted_z2,sorted_mat = zip(*sorted_mat) return np.array(sorted_mat).T, sorted_z1, sorted_z2
%matplotlib inline import pylab as plt import seaborn import numpy as np X = (data.get_values()[1:]=="True") plot_matrix(*sort_by_cluster(X, np.argmax(z1, axis=1), np.argmax(z2, axis=1)))

ここには載せないけど、以下のようにユーザ名とクラスタを確認した。
概ねどっちも大丈夫に思うけれど、z1(フォローする人でクラスタリング)のほうは直感とやや異なる部分もあった。
temp = list(zip(uname, np.argmax(z1, axis=1))) temp.sort(key=lambda x:x[1]) #temp
temp = list(zip(uname, np.argmax(z2, axis=1))) temp.sort(key=lambda x:x[1]) #temp
おわりに
PythonでSBMを実装した。
概ねできていると思うが、収束しているのか、そもそもサンプルがちゃんと独立になっているかなど不安な面もいくつかある。
自分で実装を書けばStan(もしくはEdward)でも書けるようになるかなと思ったけど、今のところよくわからない。
事後確率まで書いてサンプリングだけ頼っても意味ない気がするし。
今回はギリシャ文字とか使いまくってプログラムを書いてみた。
数式とできるだけ同じように書くとわかりやすくていいと思う。
EmacsでTeX書式で入力できるようにしたり、半角にしたりすると、特に違和感もなかった。
ただ、デバッグしたり、別の環境でソースコードを見るときは大変かもしれない。
またpythonだと₁とか∇とかを使えないので、z1とか統一できない部分もいくつかあって微妙だった。