概要
関係データ学習の後半のうち,行列分解を実装した.
lossが下がっているので,たぶん合っていると思うが,分解したあとのベクトルを見るとよくわからない.
はじめに
以前こんなものをやった.
ksknw.hatenablog.com
前半部分は終わったので(SBMのことは置いといて),後半部分の実装をやる.
今回は行列分解を実装する.
www.kspub.co.jp
MovieLensデータ
MovieLens 100K Dataset | GroupLens
これといって分解したい行列データもなかったので,本に書いてあったデータを使うことにする.
MovieLensにはユーザが映画を評価したデータが含まれている.
データを作成した時期によってデータのサイズが違うらしいが,今回は一番小さいサイズのデータを使う.
このデータには943人のユーザの映画の評価(1から5)が含まれている.
映画は1682本で各ユーザはこれらのうち少なくとも5本を評価している.
とりあえずデータを見てプロットする.
%matplotlib inline
import pylab as plt
import seaborn
import numpy as np
import pandas as pd
data = pd.read_csv("./ml-100k/u.data", names=["uid", "mid", "rating", "timestamp"], sep="\t")
data
|
uid |
mid |
rating |
timestamp |
| 0 |
196 |
242 |
3 |
881250949 |
| 1 |
186 |
302 |
3 |
891717742 |
| 2 |
22 |
377 |
1 |
878887116 |
| 3 |
244 |
51 |
2 |
880606923 |
| 4 |
166 |
346 |
1 |
886397596 |
| 29 |
160 |
234 |
5 |
876861185 |
| ... |
... |
... |
... |
... |
| 99995 |
880 |
476 |
3 |
880175444 |
| 99996 |
716 |
204 |
5 |
879795543 |
| 99997 |
276 |
1090 |
1 |
874795795 |
| 99998 |
13 |
225 |
2 |
882399156 |
| 99999 |
12 |
203 |
3 |
879959583 |
100000 rows × 4 columns
X = np.zeros((data["uid"].max(), data["mid"].max()))
for i,item in data.iterrows():
X[item["uid"]-1, item["mid"]-1] = item["rating"]
f, ax = plt.subplots(figsize=(7, 7))
import numpy.ma as ma
masked = ma.masked_where((X==0),X)
cmap = plt.cm.jet
cmap.set_bad('white',1.)
plt.pcolormesh(masked, cmap=cmap)
plt.xlabel("movie id")
plt.ylabel("user id")
plt.show()
f, ax = plt.subplots(figsize=(7, 7))
plt.pcolormesh(masked[:100, :100], cmap=cmap)
plt.xlabel("movie id")
plt.ylabel("user id")
plt.show()


人気の映画やよく映画を見ている人などがなんとなくわかる.
多くの人が好む映画,やたら低評価ばかりつけるユーザなど色々いる.
なんらかソートされていることもわかる.
また大半は欠損データである.
まずは,(練習なので)欠損データは0とレーティングされているとして行列分解する.
その後,欠損値を除外したり,補完をするアルゴリズムを実装する.
また,本に載っているアルゴリズムでは収束判定をしていたが省略する.
import copy
original_X = copy.deepcopy(X)
f, ax = plt.subplots(figsize=(7, 7))
plt.pcolormesh(X[:100, :100], cmap=cmap)

1次交互勾配降下法
正則化つきの行列分解は特異値分解では求まらないので,勾配法を用いる必要がある.
それぞれ5つのベクトルでユーザ,映画が表現できると考えて,行列分解する.
動けばいいやの精神で書いているので,行列の計算はあれな感じだけど許してほしい.
というかnumpy.matとarray関連の正しい使い方がわからない.
1次交互勾配法は不安定で,learning_rateを少しあげたら発散した.
nb_epoch = 100
reg = 0.01
R = 5
learning_rate = 0.001
I = X.shape[0]
J = X.shape[1]
U = np.mat(np.random.random((I, R)))
V = np.mat(np.random.random((J, R)))
dU = lambda U,V: -X*V + U * (V.T*V + reg*np.mat(np.identity(R)))
dV = lambda U,V: -X.T*U + V * (U.T*U + reg*np.mat(np.identity(R)))
error = lambda U,V: 0.5*np.sum(np.array(X-U*V.T)**2) + 0.5*reg*np.sum( np.sum(np.array(U)**2,axis=0) + np.sum(np.array(V)**2,axis=0))
err_hist = []
for i in range(nb_epoch):
e = error(U,V)
U -= learning_rate * dU(U,V)
V -= learning_rate * dV(U,V)
err_hist.append(e)
def hist_plot(err_hist):
plt.plot(err_hist)
plt.text(len(err_hist) -5, err_hist[-1]+500 ,"%.1f"%err_hist[-1])
hist_plot(err_hist)

predict = np.array(U*V.T)
np.max(predict), np.min(predict)
(10.693971705841749, -2.4352853936451466)
def compare_plot(X, predict, cmap=plt.cm.Blues, vmin=None, vmax=None):
f, ax = plt.subplots(figsize=(14, 7))
plt.subplot(121)
plt.pcolormesh(X[:100, :100], cmap=cmap, vmin=vmin, vmax=vmax)
plt.xlabel("movie id")
plt.ylabel("user id")
plt.subplot(122)
plt.pcolormesh(predict[:100, :100], cmap=cmap, vmin=vmin, vmax=vmax)
plt.xlabel("movie id")
plt.ylabel("user id")
plt.show()
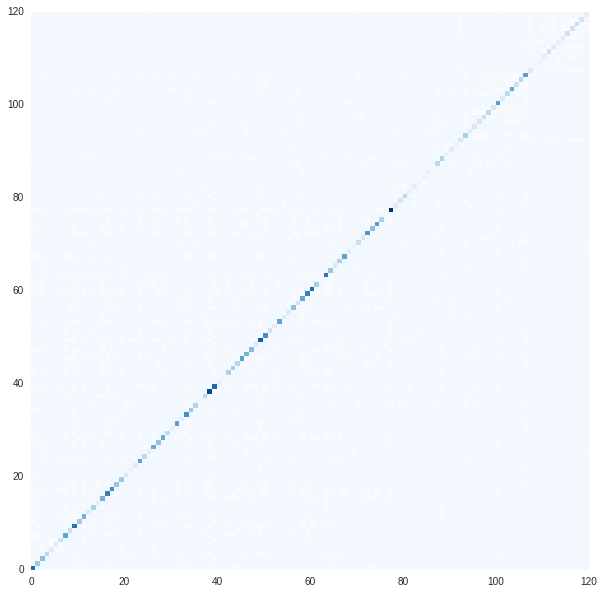
compare_plot(X, predict)

最大値,最小値がずれているのでJetだと見にくかったので,青だけでプロットしている.
なんとなく出来ている気がする.
擬似2次交互勾配法
1次勾配だけだと収束が不安定で学習率をあまり大きくすることができず,収束が遅い.
2次勾配を使うことで収束を早くすることができる.
learning_rate = 1.
I = X.shape[0]
J = X.shape[1]
U = np.mat(np.random.random((I, R)))
V = np.mat(np.random.random((J, R)))
err_hist2 = []
for i in range(nb_epoch):
e = error(U,V)
U = (1-learning_rate)*U + learning_rate * X*V*((V.T*V+reg*np.mat(np.identity(R))).I)
V = (1-learning_rate)*V + learning_rate * X.T*U*((U.T*U+reg*np.mat(np.identity(R))).I)
err_hist2.append(e)
hist_plot(err_hist)
hist_plot(err_hist2)

predict = np.array(U*V.T)
np.max(predict), np.min(predict)
(10.414641991236321, -2.2410733139172789)
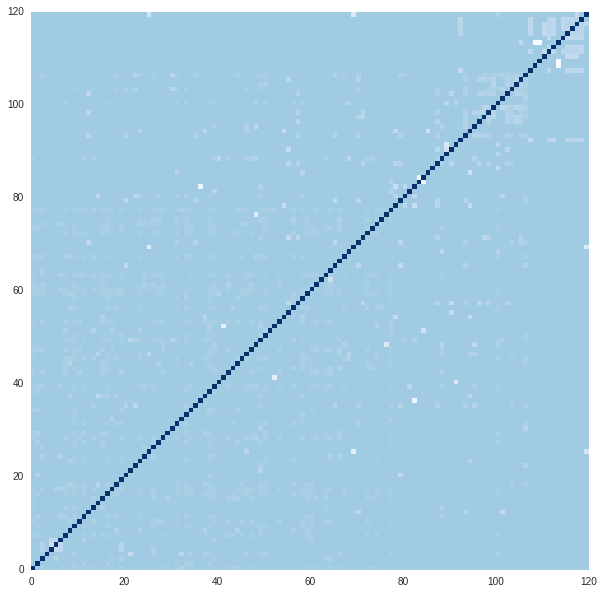
compare_plot(X, predict)

学習率を1にしても発散せず,1次勾配(青)よりも2次勾配(緑)を利用したものは早く収束するようになった.
制約付きの最適化
ここまでの実装では,推定した評価値が負の値になっている.
U,Vに制約をつけることで0以上の値にすることができる. (評価値は1から5までなので本当はもう少し制約しないといけないがどうするのかはわからない.)
具体的には,負の値をとった要素を0に射影しながら最適化する.
learning_rate = 1.
I = X.shape[0]
J = X.shape[1]
U = np.mat(np.random.random((I, R)))
V = np.mat(np.random.random((J, R)))
err_hist_con = []
for i in range(nb_epoch):
e = error(U,V)
U = (1-learning_rate)*U + learning_rate * X*V*((V.T*V+reg*np.mat(np.identity(R))).I)
U = np.maximum(U, 0, U)
V = (1-learning_rate)*V + learning_rate * X.T*U*((U.T*U+reg*np.mat(np.identity(R))).I)
V = np.maximum(V, 0, V)
err_hist_con.append(e)
hist_plot(err_hist_con)

predict = np.array(U*V.T)
np.max(predict), np.min(predict)
(8.9972633401464464, 0.0)
ちゃんと0以上に制約されている.
compare_plot(X, predict)

欠損値をちゃんとやる
ここまでは欠損値を全て0だと仮定して実装した.
以下では欠損値を除外して行列分解する方法と補完する方法を行う.
除外する方法
lossの計算およびU,Vの勾配の計算のときに,欠損した値は利用しない.
実装が楽なので1次勾配を利用する方法で実装した.
M = np.array((X!=0)*1)
I = X.shape[0]
J = X.shape[1]
U = np.mat(np.abs(np.random.random((I, R))))
V = np.mat(np.abs(np.random.random((J, R))))
dU = lambda U,V: -(np.array(X - U*V.T)*M)*V + reg*U
dV = lambda U,V: -(np.array(X - U*V.T)*M).T*U + reg*V
error = lambda U,V: 0.5*np.sum((np.array(X-U*V.T)*M)**2) + 0.5*reg*np.sum( np.sum(np.array(U)**2,axis=0) + np.sum(np.array(V)**2,axis=0))
err_hist = []
for i in range(nb_epoch):
e = error(U,V)
U = (1-learning_rate)*U + learning_rate * X*V*((V.T*V+reg*np.mat(np.identity(R))).I)
V = (1-learning_rate)*V + learning_rate * X.T*U*((U.T*U+reg*np.mat(np.identity(R))).I)
err_hist.append(e)
hist_plot(err_hist)

predict = np.array(U*V.T)
np.max(predict), np.min(predict)
(10.414841938424392, -2.2413625198086238)
import numpy.ma as ma
masked_predict = ma.masked_where((X==0),predict)
compare_plot(masked[:100, :100], masked_predict, cmap=cmap, vmin=1, vmax=5)
compare_plot(masked[:100, :100], predict, cmap=cmap, vmin=1, vmax=5)


全体的に評価が低めな感じになってしまっている.
また,欠損していた部分も低めの値で埋まっている感じ.
補完する方法
こちらの方法では欠損値を毎回,予測値で補完する.
欠損値を除外する方法と等価らしいので,こちらは2次勾配を使って,制約もつける方法で実装する.
M_hat = np.array((X==0)*1)
I = X.shape[0]
J = X.shape[1]
U = np.mat(np.abs(np.random.random((I, R))))
V = np.mat(np.abs(np.random.random((J, R))))
error = lambda U,V: 0.5*np.sum((np.array(X-U*V.T)*M)**2) + 0.5*reg*np.sum( np.sum(np.array(U)**2,axis=0) + np.sum(np.array(V)**2,axis=0))
err_hist_comp = []
X_hat = X + np.mat(np.array(U*V.T)*M_hat)
for i in range(nb_epoch):
e = error(U,V)
U = (1-learning_rate)*U + learning_rate * X_hat*V*((V.T*V+reg*np.mat(np.identity(R))).I)
U = np.maximum(U, 0, U)
X_hat = X + np.mat(np.array(U*V.T)*M_hat)
V = (1-learning_rate)*V + learning_rate * X_hat.T*U*((U.T*U+reg*np.mat(np.identity(R))).I)
V = np.maximum(V, 0, V)
X_hat = X + np.mat(np.array(U*V.T)*M_hat)
err_hist_comp.append(e)
hist_plot(err_hist_comp)

predict = np.array(U*V.T)
np.max(predict), np.min(predict)
(7.7494405681324903, 0.0082254500224862269)
import numpy.ma as ma
masked_predict = ma.masked_where((X==0),predict)
compare_plot(masked[:100, :100], masked_predict, cmap=cmap, vmin=1, vmax=5)
compare_plot(masked[:100, :100], predict, cmap=cmap, vmin=1, vmax=5)


だいたいできているような気がする.
ベクトルをみる
行列分解で得られたU,Vでは近いユーザもしくは映画が近いベクトルで表現される.
MovieLensには評価だけでなく,どんな映画かやどんなユーザかといった情報も含まれている.
これらとU,Vを比較することで,正しい行列分解ができたかを評価する.
普段あまり映画を見ないのと,古い映画のデータしかないので,Toy StoryとGolden Eyeぐらいしかわからなかった.
ユーザの属性ならなんとなくわかるので,そちらを使う.
ユーザの情報は以下.
user_info = pd.read_csv('ml-100k/u.user', sep='|',
encoding='latin-1',header=None)
user_info
|
0 |
1 |
2 |
3 |
4 |
| 0 |
1 |
24 |
M |
technician |
85711 |
| 1 |
2 |
53 |
F |
other |
94043 |
| 2 |
3 |
23 |
M |
writer |
32067 |
| 3 |
4 |
24 |
M |
technician |
43537 |
| ... |
... |
... |
... |
... |
... |
| 937 |
938 |
38 |
F |
technician |
55038 |
| 938 |
939 |
26 |
F |
student |
33319 |
| 939 |
940 |
32 |
M |
administrator |
02215 |
| 940 |
941 |
20 |
M |
student |
97229 |
| 941 |
942 |
48 |
F |
librarian |
78209 |
| 942 |
943 |
22 |
M |
student |
77841 |
943 rows × 5 columns
職業がわかりやすそうなので,プロットしてみる.
from sklearn.manifold import TSNE
tsne = TSNE()
tsned = tsne.fit_transform(U)
plt.scatter(tsned[:,0], tsned[:,1], c=(user_info[3]=="scientist"), cmap=plt.cm.Accent, marker=".")
plt.show()
plt.scatter(tsned[:,0], tsned[:,1], c=(user_info[3]=="engineer"), cmap=plt.cm.Accent, marker=".")
plt.show()
plt.scatter(tsned[:,0], tsned[:,1], c=(user_info[3]=="student"), cmap=plt.cm.Accent, marker=".")
plt.show()



そうでもない感じだったので,うまくいっているのかよくわからない.
まとめ
勉強のために行列分解の実装をした.
収束はしたけど,うまくいっているのかはよくわからない.
参考

。これと、πの合計が1になるように、適当に制約を加えて、以下のように誤差関数を定義した。